12月8日,来自深势科技的团队推出了蛋白质结构预测工具Uni-Fold,并开源训练、推理代码。Uni-Fold成功复现了曾引起生物学界轰动的Alphafold2的全规模训练,相应解决方案已集成至深势科技自主研发的药物设计平台Hermite,供广大用户测试使用。Uni-Fold克服了Alphafold2未开源训练代码、硬件支持单一、模型不可商用等局限性,在训练与推理环节进行了GPU上的适配、性能优化、功能完善等工作,为更多人参与推动领域进一步发展提供了基础。

图1 Uni-Fold预测的蛋白结构
蛋白质是组成生命体的重要物质基础。不同的蛋白质通过折叠形成不同的三维结构,执行多种多样的生理功能。从氨基酸序列到对应蛋白质三维结构的预测问题被认为是生物学领域最具有挑战性的问题之一。在过去几十年的研究中,科学家们为探测蛋白质结构发展出三大实验手段:X射线晶体学、核磁共振和冷冻电镜。但实验方法成本高、周期长。目前人类已知有数十亿氨基酸序列,但还原出结构的只有十几万。生物学发展因此颇受掣肘。
近年来AI的发展为这一问题带来了新的可能。在2020年的国际蛋白质结构预测顶级竞赛CASP14(Critical Assessment of protein Structure Prediction 14)上,Google DeepMind推出的AlphaFold2预测的大部分结构准确度均与实验足够接近,远超此前所有方法。这一结果也标志着蛋白质单体的结构问题在一定意义上得到了解决。
不过,尽管DeepMind在半年后开源了AlphaFold2模型的推理代码,其训练代码并未被公布,且模型不可商用。研究者难以通过训练或调整模型进一步提升AlphaFold2的表现,或迁移至更多应用场景,如蛋白质复合物结构预测、蛋白质与小分子相互作用、与电镜实验结合等。此外,该代码基于Google自研的可微分计算框架JAX及TPU硬件平台开发,高度依赖Google生态系统。
Uni-Fold打破了这些限制。目前已开源的Uni-Fold训练代码,经过MPI并行、数据异步读取、混合精度训练等优化工作,支持高效GPU训练。基于优化后的训练代码,深势科技团队实现在128块NVIDIA GPU上进行AlphaFold2模型的全规模训练,并将持续优化所得模型。Uni-Fold的推理代码更加轻量、高效,在相同硬件环境下,可获得与公布的Alphafold2代码相比2-3倍的效率提升。
在相同的测试条件下,Uni-Fold的预测精度超越了华盛顿大学蛋白设计研究所David Baker教授课题组研发的RoseTTAFold,与DeepMind官方发布的AlphaFold2模型接近。深势科技团队使用Uni-Fold对CASP14蛋白预测任务中的序列数据集进行了直接测试,平均Cα-lDDT达到82.6。可以说,Uni-Fold是“训练公开版的AlphaFold2”,而且硬件适配与效率更加优化。
中科院院士、北京大学教授、深势科技首席科学顾问鄂维南评价道:“尽管DeepMind开源了推理代码,但模型的训练技术才是核心竞争力。在资源比较匮乏的情况下,深势科技团队在较短时间内实现模型训练、开源训练代码,这是很不容易的。深势科技在物理模型方面具备丰富的经验,Uni-Fold进一步弥补了团队在数据驱动方面的短板。下一步,物理模型和数据驱动结合的解决方案将为蛋白结构和药物设计行业提供更好的基础。”
深势科技也对Uni-Fold进行了进一步的开发和优化,集成于旗下自主研发的一站式计算辅助药物设计平台Hermite中,供用户使用。Hermite是根植于云计算的临床前药物设计全流程计算平台,致力于为药物科研工作者提供数据、算法、算力三位一体的一站式解决方案。在Uni-Fold前,Hermite上已集成多个重量级功能。例如基于Uni-Fold等方法得到蛋白结构后进行蛋白的动力学模拟、结构精修或环区优化的RiD模块;在药物优化阶段比较不同分子与蛋白靶点结合活性差别的Uni-FEP模块,等等。
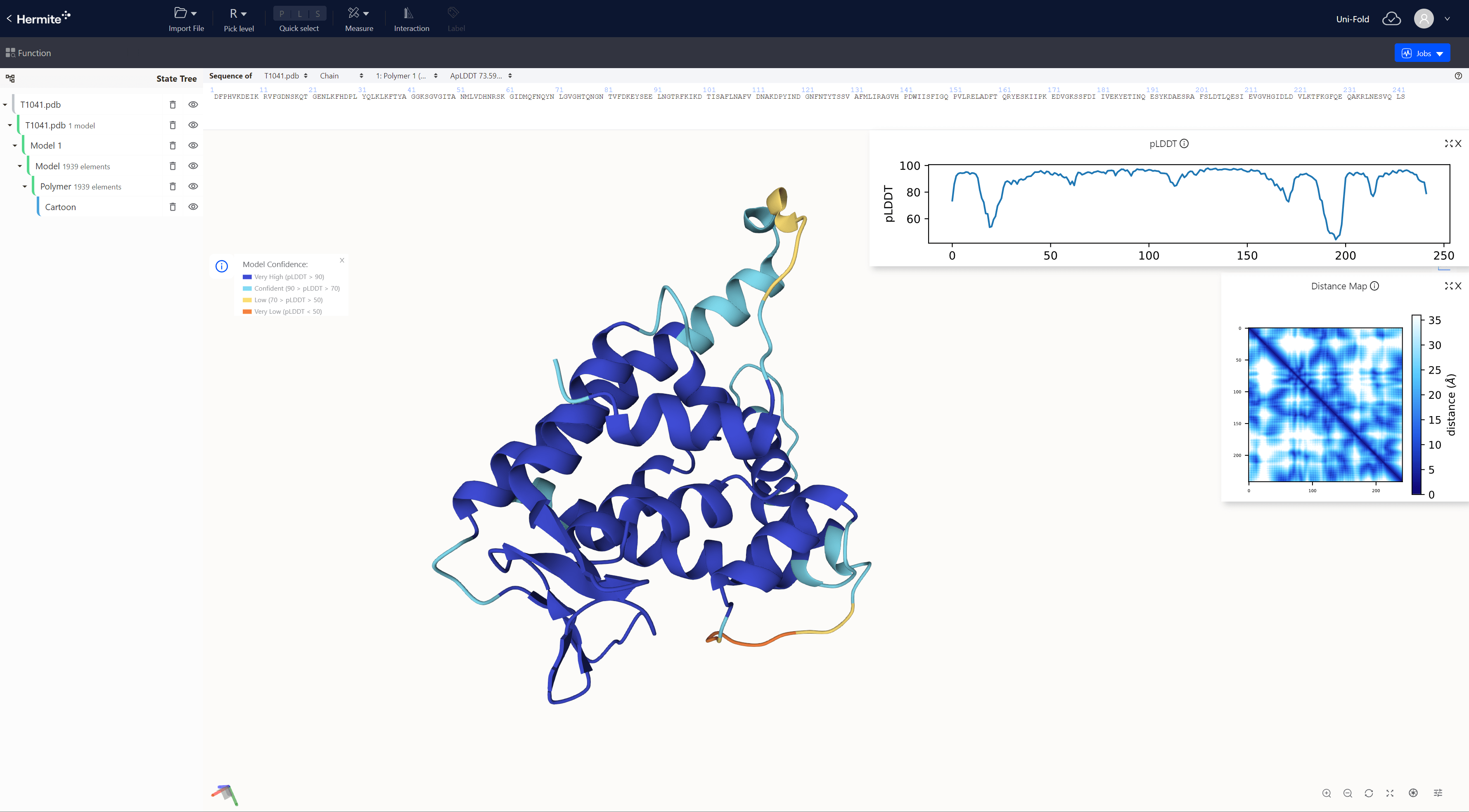
图2 Uni-Fold在深势科技Hermite药物设计平台上的使用
在药物设计领域,结合Uni-Fold、RiD等AI辅助计算模拟的算法,以及算法与冷冻电镜等实验手段的结合,深势科技正在打造出一套模拟—AI—实验三者驱动的解决方案,赋能药物设计行业,加速从业者的药物研发进程。
芝加哥丰田计算技术研究所的许锦波教授主攻机器学习、优化方法和计算生物学,也是深度学习预测蛋白质结构的先驱。他评价道:“针对AlphaFold未公开训练代码的局限性,深势科技敢于打破现状,率先公布训练代码。Uni-Fold的出现,让每个研究者都可以训练自己的模型。这无疑将进一步推动技术的发展。对整个蛋白质折叠领域来说,都是令人激动的消息。”

 高层
高层 访谈
访谈

 观点
观点

